NCBI_GEOからのNGSデータの取得方法 - suimye/NGS_handson2015 GitHub Wiki
2. ChIP-seqデータのGEOからの取得方法
論文からGSEナンバーをcheckして、GEOから検索します。今回は、SoufiらのiPS細胞に関する2012年の論文(Facilitators and Impediments of the Pluripotency Reprogramming Factors' Initial Engagement with the Genome)からOct4のChIP-seqデータに興味をもったと仮定して、データを取得してみましょう。
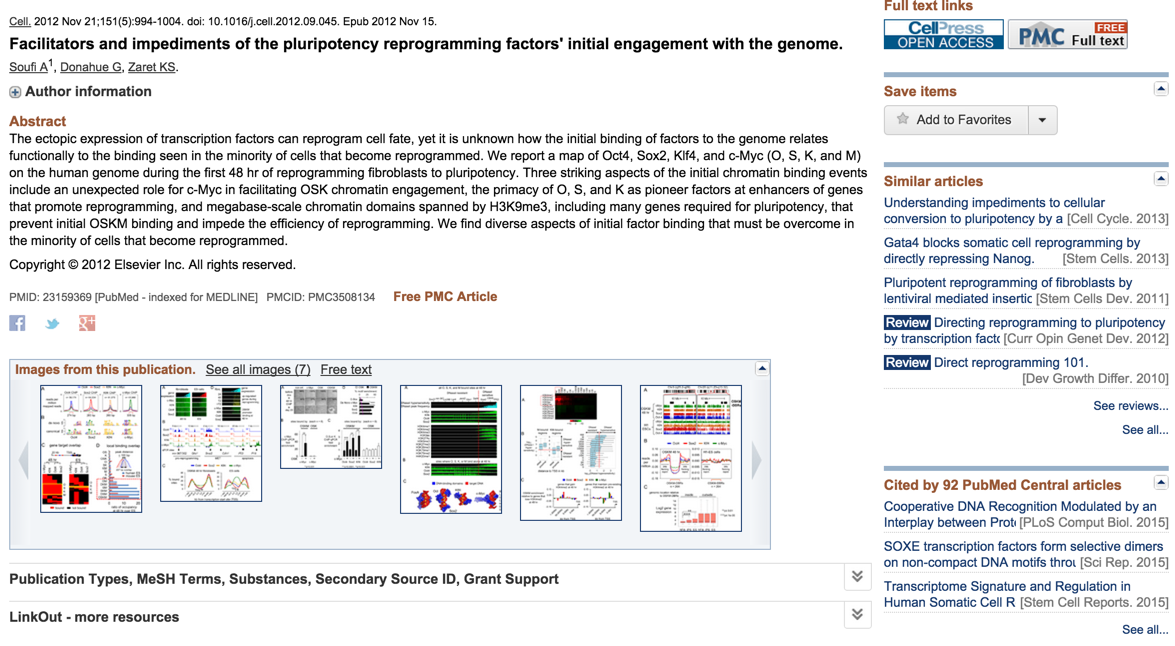 文献の中には、GSEナンバーが書いてあります。
文献の中には、GSEナンバーが書いてあります。
 このように、論文に記載されているGSEナンバーを元に必要なデータをたどる方法は、再解析からどのような結果が期待できるのかを、あらかじめ文献をしっかり読んだ上で推測することができるので、お勧めの方法です。
このように、論文に記載されているGSEナンバーを元に必要なデータをたどる方法は、再解析からどのような結果が期待できるのかを、あらかじめ文献をしっかり読んだ上で推測することができるので、お勧めの方法です。
###GEOでの検索 では、NCBIの遺伝子発現オムニバスGEO, GEOのデータセット検索にアクセスしてみましょう。
http://www.ncbi.nlm.nih.gov/gds/
検索対象がGEO Datasetsになっていることを確認して、検索キーワードをiPS Oct4などで検索してみましょう。
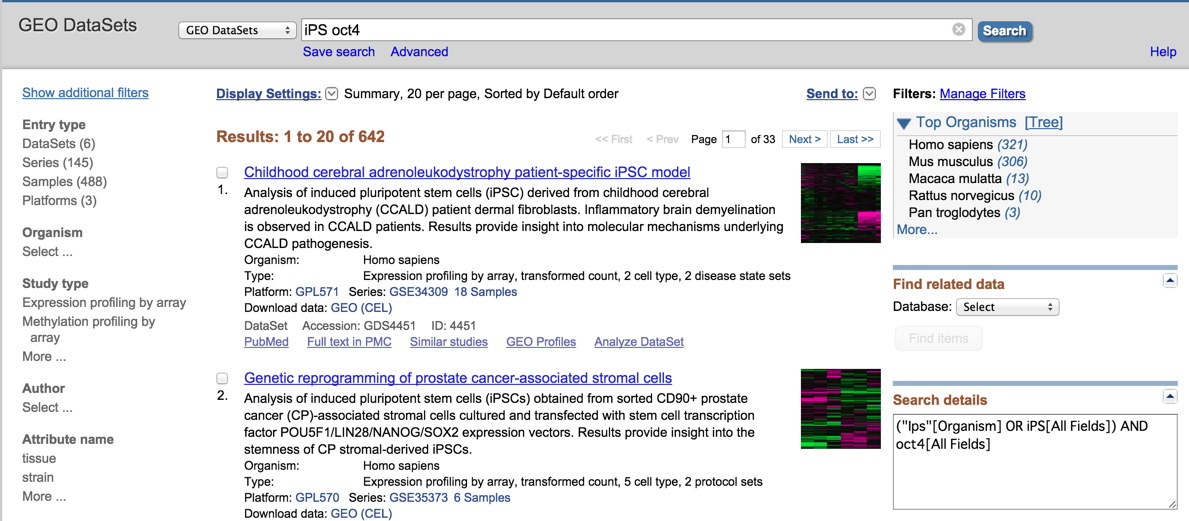
ところが、目的のSoufiらの論文に1ページ目ではたどり着けません。2012年の論文は現在から既に5年が経過しているために、押し流されているので、やはりもっと限定的なキーワードがよいということです。では次に、GSEナンバーに基づいてみましょう。
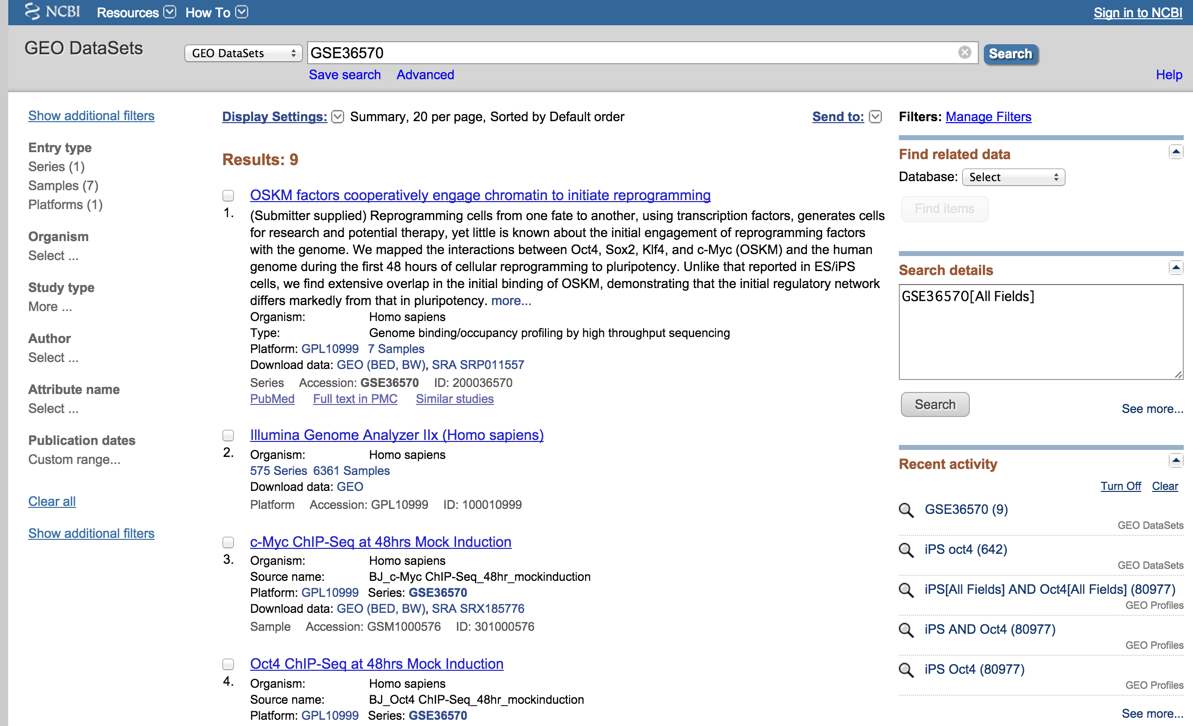
topヒットをクリックしてください。
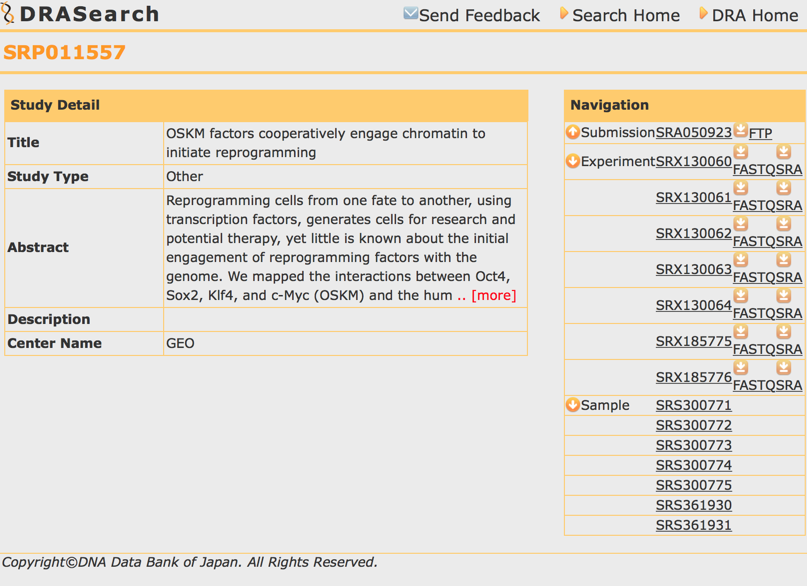
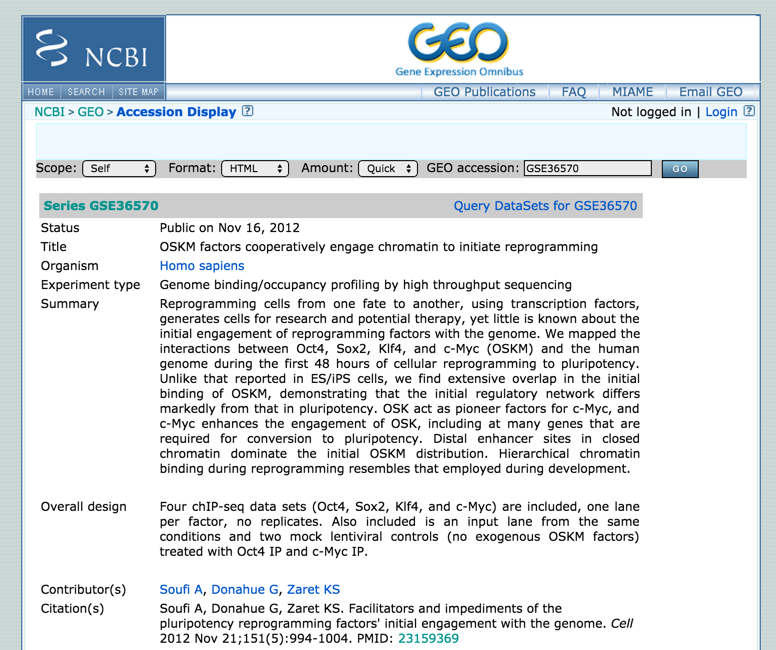 GSEはSeriesAccessionなので、その論文の著者がGEOにシリーズとして登録しているデータのデータの概要(アブストラクト)を出してくれています。再解析をするときは、最低でもこの内容に目を通すことが重要です。
GSEはSeriesAccessionなので、その論文の著者がGEOにシリーズとして登録しているデータのデータの概要(アブストラクト)を出してくれています。再解析をするときは、最低でもこの内容に目を通すことが重要です。
従って、iPS細胞のOct4に関連するChIP-seqデータが欲しいという漠然とした検索対象でGEOから探すよりも、論文を読んで、研究デザインに基づいて情報を抽出する方が確実な再解析できると思います。もちろん、データ先導的な解析をしたい場合は、前者のような方法も有効ですがその場合はもう少し自動的にデータをフィルタリングするなどの工夫を行って、大量のデータを取得する方が望ましいでしょう。
GEOからのダウンロード
ここではGEOの使い方をお話しいたしますが、実際にダウンロードして解析するのは、他のテストデータになります。NGSデータなどのリポジトリのデータは、
GSEナンバー => GSMナンバー => SRX => SRRナンバー (NGSデータの固有番号)
という、ある研究 -> 個々のデータのGEO登録ナンバー =>NGSファイルという階層で構成されています。
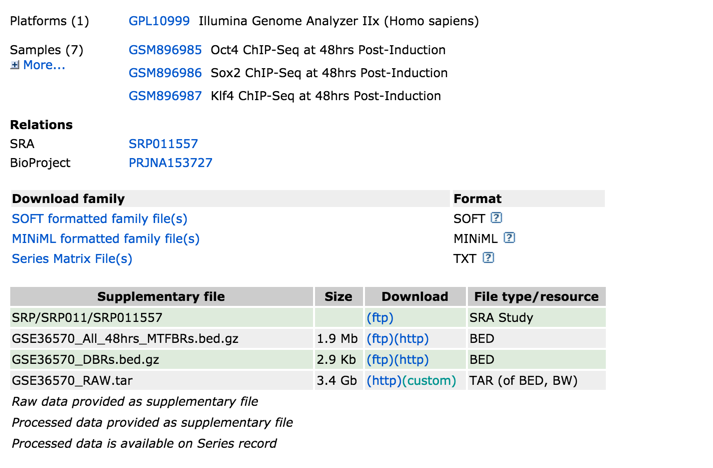
URL下段には、Download familyや、Supplemental filesとして色々なデータが登録されています。 先にsupplemental filesをみてみると、
- SRP/SRP011/SRP011557 (ftp) SRA Study
- GSE36570_All_48hrs_MTFBRs.bed.gz 1.9 Mb (ftp)(http) BED
- GSE36570_DBRs.bed.gz 2.9 Kb (ftp)(http) BED
- GSE36570_RAW.tar 3.4 Gb (http)(custom)TAR (of BED, BW)
これらのファイルは、生データ(NGSデータ)をグループとしてftpに置いてあるものや、NGS解析の過程でなんらかの処理がなされたファイル(processed data、ここではBEDファイル)が登録されています。Processed dataは、どんなデータ形式をGEOに置くかは特に取り決めがあるわけではないデータで、登録者が論文に報告する際に要求されたデータや解析に使用したデータです。つまりエントリーごとに、置かれているデータもそのファイル形式も違います。
1サンプルのダウンロードの場合はDownload familyからが便利です。この項目には、サンプル毎にリンクとそのサンプルの研究における名前が書かれてあり、大変便利です。GSMナンバーをクリックし、次のページの下段にあるSRXナンバーをクリックすればダウンロードすることができます。
捕捉: その他のダウンロード方法
- wgetを利用する
wget というコマンドがインストールされていれば、次のコマンドでダウンロードすることができます。SRRナンバーのURLをコピー (wget利用する、NCBI toolkitを使う)しておき、次のようにコマンドを実行します。
ダウンロード後に、sraファイルを解凍するには。
fastq-dump SRR445816.sra
- SRRナンバーを使って、fastq-dump -Aを利用する (NCBI toolkitを使う)
NCBI toolkitを使ったダウンロード方法です。sraファイルをダウンロードしつつ解凍もしてくれます。wgetを使うよりも便利ですね。
fastq-dump -A SRR445816.sra
GEO以外のデータベース
DRA
- fastqでデータが保持されているので、sraからの変換が要らない。
- GEOのsraデータが破損している場合などにこちらのfastqファイルは破損していなかったなどの経験あり。
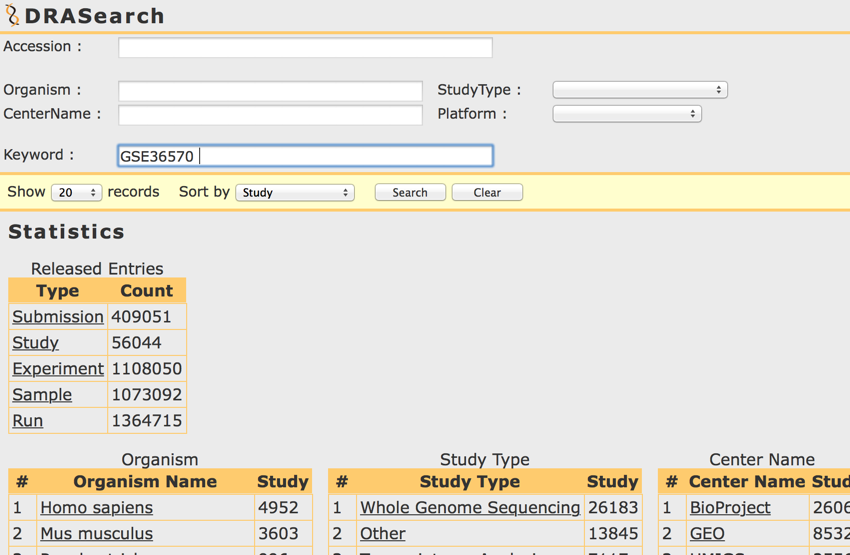
- GSEナンバーを入れるときは、keywordのところへ
- fastqが保持されているので解析しやすい。
ENA
- DRAと同様にfastqファイルを保持してくれている。
- DRAがメンテナンス中のときはこちらを使う事も。