Knime_podstawy - filipsPL/CADD-PW GitHub Wiki
- Troubleshooting
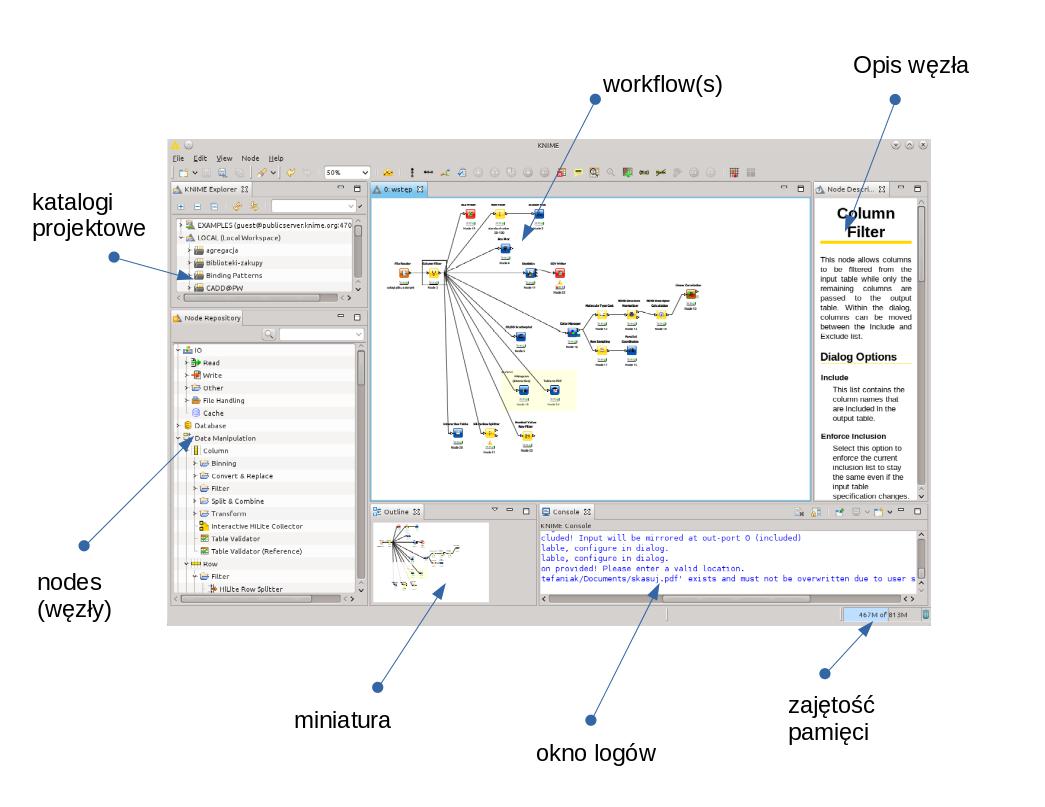
- Co jest czym w KNIME
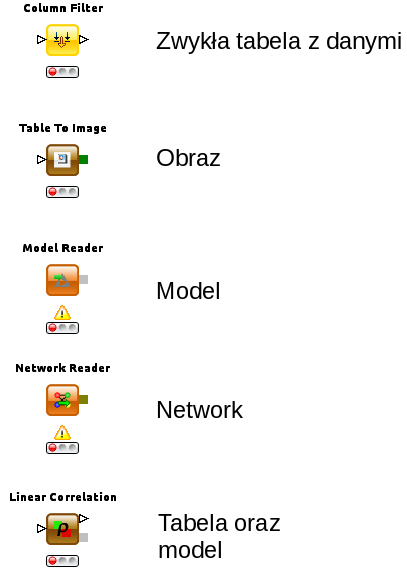
- Węzły - Nodes
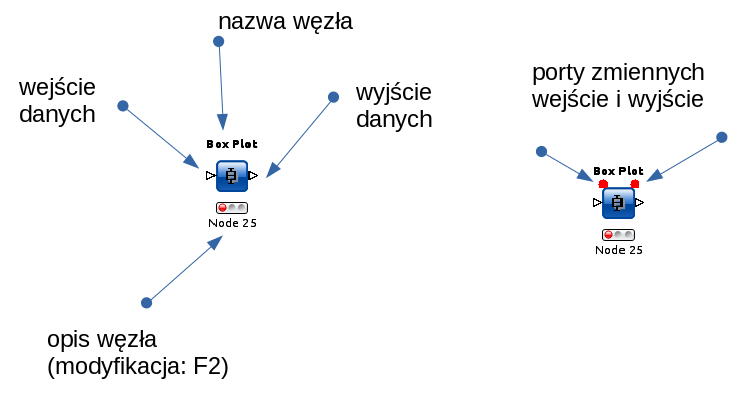
- Wejścia - wyjścia nodów
- Opcje węzła
- Skróty klawiszowe
- Opis niektórych węzłów
Proszę pobrać na dysk plik z danymi (wartości IC50 dla enzymu JAK2)
czyli często spotykane problemy
File -> switch workspace; wybrać swój katalog lub stworzyć nowy
Gdy w tabeli nie pojawiają się struktury chemiczne: prawy przycisk myszy na nagłówek, zmienić render -> marvin lub indigo
Należy zapisać workflow, zamknąć go i powtórnie otworzyć.
Kliknięcie prawym przyciskiem myszy na węźle.
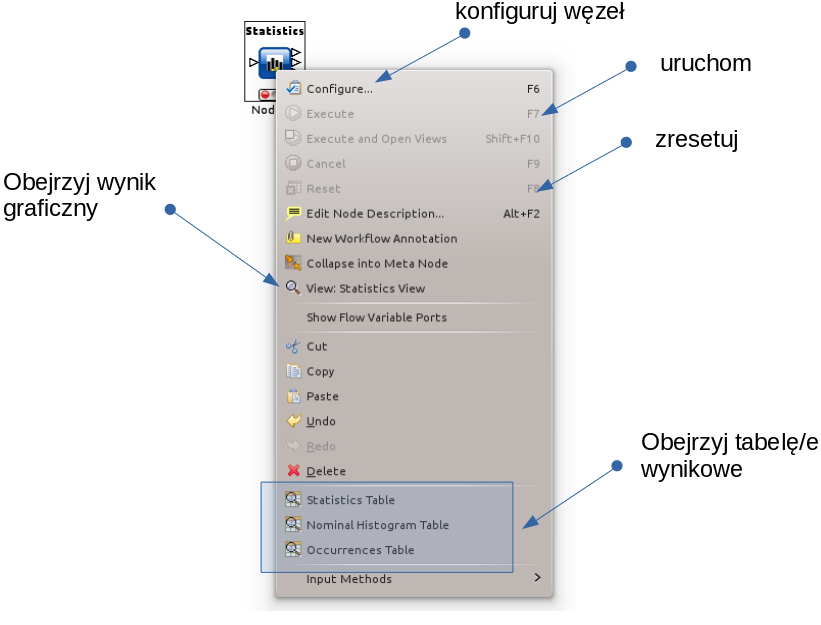
F6 - konfiguruj węzeł
F7 - uruchom węzeł (i wszystkie poprzedzające)
F2 - edytuj opis węzła
Ctrl + S - zapisz workflow
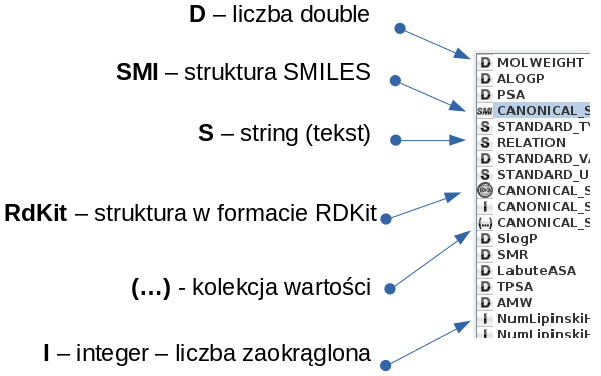
Niektóre możliwe typy zmiennych / kolumn:
| Węzeł | Opis |
|---|---|
| file reader | czyta dowolny plik tekstowy |
| row filter | filtrowanie wierszy (wg wartości, zakresu... |
| column filter | Wywalenie niektórych kolumn |
| csv writer | zapisuje tabelę jako plik tekstowy csv (do odczytania np w excelu) |
| xls writer | zapisuje tabelę jako arkusz excela |
| table to PDF | zapisuje tabelę do pdf'a |
| statistics | liczy różne parametry statystyczne dla wybranych danych |
| nominal value row filter | filtruje dane wg zadanych wartości (np kolor = zielony i czerwony) |
| row splitter | dzieli wejściową tabelę na dwie wg zadanych kryteriów |
| numeric binner | dzieli wartości liczbowe na kategorie |
| GroupBy | grupuje dane wejściowe wg zadanych kryteriów |
| Linear Correlation | Korelacja liniowa - kolorowa macierz |
| Row Sampling | Zmniejszenie liczby wierszy poprzez wybór: pierwszych z tabeli / przypadkowej reprezentacji n wierszy |
| Interactive table | Tabela umożliwiająca wybór wierszy i ich zakreślenie (HiLite) |
| HiLite Row Splitter | Podział danych wejściowych na te zakreślone (HiLite) i pozostałe |
| Color Manager | Pokolorowanie rekordów wg wybranych wartości liczbowych (np MW, TPSA, aktywności...) |
| Numeric binner | Kategoryzacja wartości liczbowych wg zadanych progów (np na aktywne i nieaktywne itp) |
| Pareto ranking | Próba znalezienia kompromisu między kilkoma zmiennymi. Im wyższe miejsce w rankingu tym lepiej |
| GroupBy | Grupowanie po jakiejś kategorii |
| Węzeł | Opis |
|---|---|
| Molecule type cast | Zamienia dane tekstowe (np ciąg SMILES) na dane strukturalne |
| Structure normalizer | Standardyzacja / normalizacja struktur chemicznych |
| Descriptor calculation | Liczenie parametrów cząsteczek (np MW, logP, TPSA...) |
| Murcko Scaffolds | Wyznacza scaffoldy (szkielety, rusztowania) związków |
| MCS | Wyznacza Maximum Common Substructure - maksymalną wspólną podstrukturę, wspólną dla wszystkich związków |
| Marvin Sketch | Rysowanie struktur chemicznych |
| RDKit Molecule Substructure Filter | Wyszukuje podstruktur w tabeli wejściowej |
| RDKit fingerprint | Liczy fingerprint ("00010100010110001...")dla struktur w tabeli wejściowej |
| Fingerprint Similarity | Podobieństwo fingerprintów = podobieństwo struktur chemicznych |
| RDKit Diversity Picker | Wybiera różnorodny (zdywersyfikowany) zbiór struktur |
| Similarity viewer | Wyświetla heatmap (mapę ciepła) obrazującą zróżnicowanie struktur w tabeli wejściowej. Musi być poprzedzony węzłami: liczenia fingerprintów i Distance Matrix Calculate |