遗传算法&TSP - ZYL-Harry/Mathematical_Modeling_Algorithms GitHub Wiki
遗传算法&TSP的问题介绍
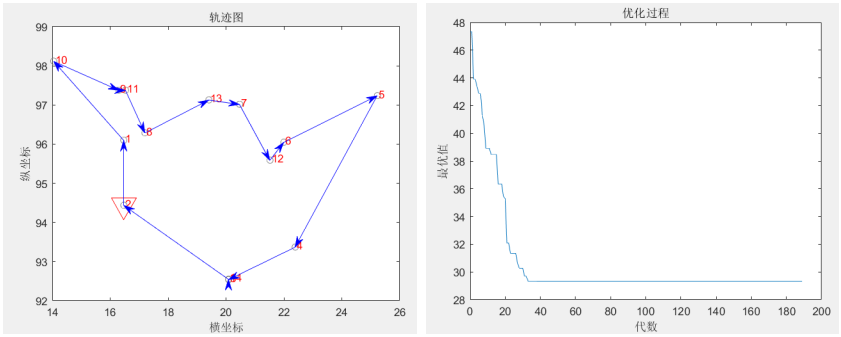
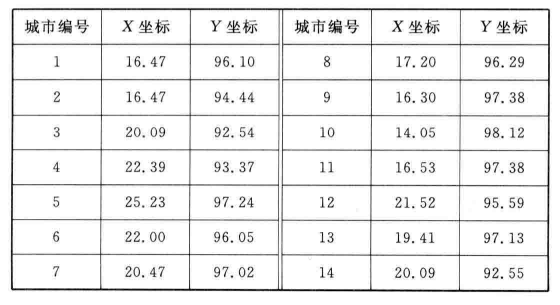
本案例以14个城市为例,假定14个城市的位置坐标如表所列,寻找出一条最短的遍历14个城市的路径
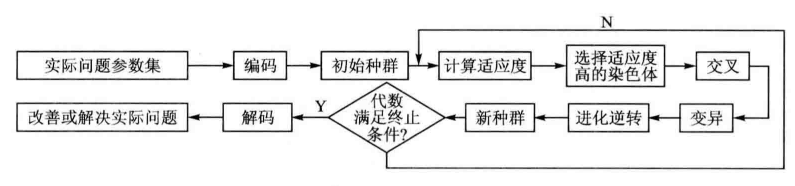
算法步骤
算法流程图
算法步骤
-
编码
采用整数排列编码方法。对于 n 个城市的 TSP 问题,染色体分为n段,其中每一段为对应城市的编号,如对 10 个城市的 TSP 问题{1,2,3,4,5,6,7,8,9,10},则 11 | 10 | 2 1 4 1 5 1 6 1 8 1 7 1 9 1 3 就是一个合法的染色体。 -
种群初始化
在完成染色体编码以后,必须产生一个初始种群作为起始解,所以首先需要决定初始化种群的数目。初始化种群的数目一般根据经验得到,一般情况下种群的数量视城市规模的大小而确定,其取值在 50~200 之间浮动。 -
适应度函数
设 k1 | k2 | … | ki |…| kn |为一个采用整数编码的染色体,D_kikj为城市ki到城市kj,的距离,则该个体的适应度为
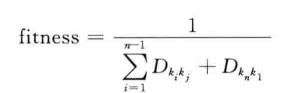
即适应度函数为恰好走遍π个城市,再回到出发城市的距离的倒数。优化的目标就是选择适应度函数值尽可能大的染色体,适应度函数值越大的染色体越优质,反之越劣质。 -
选择操作
选择操作即从旧群体中以一定概率选择个体到新群体中,个体被选中的概率跟适应度值有关,个体适应度值越大,被选中的概率越大。 -
交叉操作
采用部分映射杂交,确定交叉操作的父代,将父代样本两两分组,每组重复以下过程(假定城市数为 10):
①产生两个[1,10]区间内的随机整数r1和r2,确定两个位置,对两位置的中间数据进行交叉,如r1=4,r2=7

交叉为

②交叉后,同一个个体中有重复的城市编号,不重复的数字保留,有冲突的数字(带*位置)采用部分映射的方法消除冲突,即利用中间段的对应关系进行映射。结果为

-
变异操作
变异策略采取随机选取两个点,将其对换位置。产生两个[1,10]范围内的随机整数r1和r2,确定两个位置,将其对换位置,如r1=4,r2=7
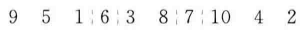
变异后为
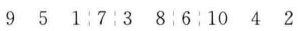
-
进化逆转操作
为改善遗传算法的局部搜索能力,在选择、交叉,变异之后引进连续多次的进化逆转操作。这里的“进化”是指逆转算子的单方向性,即只有经逆转后,适应度值有提高的才接受下来,否则逆转无效。
产生两个[1,10]区间内的随机整数r1和r2,确定两个位置,如r1=4,r2=6,算出它们之间的基因数中最大的数和最小的数,然后将它们之间的数作为向量,按两数中的大数排序至小数(即当大的数是位置靠后的数,则进行反向排序,否则正常排序)
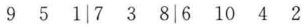
进化逆转后为
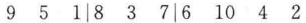
对每个个体进行交叉变异,然后代人适应度函数进行评估,选择出适应值大的个体进行下一代的交叉和变异以及进化逆转操作。循环操作:判断是否满足设定的最大遗传代数 MAXGEN,不满足则跳入适应度值的计算;否则,结束遗传操作。
MATLAB程序
1 初始化参数(包括遗传算法参数)
X =[16.47,96.10
16.47,94.44
20.09,92.54
22.39,93.37
25.23,97.24
22.00,96.05
20.47,97.02
17.20,96.29
16.30,97.38
14.05,98.12
16.53,97.38
21.52,95.59
19.41,97.13
20.09,92.55];%个城市坐标位置,可以换成load CityPosition1.mat
NIND=100; %种群大小
MAXGEN=200;
Pc=0.9; %交叉概率
Pm=0.05; %变异概率
GGAP=0.9; %代沟(Generation gap)
D=Distanse(X); %生成距离矩阵
N=size(D,1); %(34*34)
2 种群初始化
Chrom=InitPop(NIND,N);:主函数中的语句,调用InitPop函数来生成初始种群
InitPop函数function Chrom=InitPop(NIND,N) Chrom=zeros(NIND,N);%用于存储种群 for i=1:NIND Chrom(i,:)=randperm(N);%随机生成初始种群 end
3 计算每种路线的适应度
ObjV=PathLength(D,Chrom); %计算路线长度
FitnV=Fitness(ObjV); % 每种路线的适应度
Fitness函数function FitnV=Fitness(len) FitnV=1./len;
- 取每条路线的长度的倒数作为适应度,从而路线长度越短,适应度越大,染色体的个体越优
4 选择
SelCh=Select(Chrom,FitnV,GGAP);:主函数的语句,调用Select函数从旧群体选择几个个体到新群体中,这里用到的是随机普遍取样法(Stochastic Universal Sampling)
Select函数function SelCh=Select(Chrom,FitnV,GGAP) NIND=size(Chrom,1); NSel=max(floor(NIND*GGAP+.5),2); ChrIx=Sus(FitnV,NSel); SelCh=Chrom(ChrIx,:);
- 其中先得到需要选择的染色体个体个数,然后调用
Sus函数使用随机普遍取样法进行选择
Sus函数function NewChrIx = Sus(FitnV,Nsel) % Identify the population size (Nind) [Nind,ans] = size(FitnV); % Perform stochastic universal sampling cumfit = cumsum(FitnV); trials = cumfit(Nind) / Nsel * (rand + (0:Nsel-1)'); Mf = cumfit(:, ones(1, Nsel)); Mt = trials(:, ones(1, Nind))'; [NewChrIx, ans] = find(Mt < Mf & [ zeros(1, Nsel); Mf(1:Nind-1, :) ] <= Mt); % Shuffle new population [ans, shuf] = sort(rand(Nsel, 1)); NewChrIx = NewChrIx(shuf);
5 交叉
SelCh=Recombin(SelCh,Pc);:主函数中的语句,调用Recombin函数进行交叉,这里采用部分映射交叉
Recombin函数function SelCh=Recombin(SelCh,Pc) NSel=size(SelCh,1); for i=1:2:NSel-mod(NSel,2) if Pc>=rand %交叉概率Pc [SelCh(i,:),SelCh(i+1,:)]=intercross(SelCh(i,:),SelCh(i+1,:)); end end
- 先选择进行交叉的两个位置,然后调用
intercross函数进行部分映射交叉
intercross函数function [a,b]=intercross(a,b) L=length(a); r1=randsrc(1,1,[1:L]); r2=randsrc(1,1,[1:L]); if r1~=r2 a0=a;b0=b; s=min([r1,r2]); e=max([r1,r2]); for i=s:e a1=a;b1=b; a(i)=b0(i); b(i)=a0(i); x=find(a==a(i)); y=find(b==b(i)); i1=x(x~=i); i2=y(y~=i); if ~isempty(i1) a(i1)=a1(i); end if ~isempty(i2) b(i2)=b1(i); end end end
6 变异
SelCh=Mutate(SelCh,Pm);:主函数中的语句,调用Mutate函数进行变异操作,这里将染色体个体的两个元素的位置交换作为变异手段
Mutate函数function SelCh=Mutate(SelCh,Pm) [NSel,L]=size(SelCh); for i=1:NSel if Pm>=rand R=randperm(L); SelCh(i,R(1:2))=SelCh(i,R(2:-1:1)); end end
7 进化逆转操作
SelCh=Reverse(SelCh,D);:主函数中的语句,调用Reverse函数进行进化逆转操作,即产生两个基因变量区间内的随机整数,算出它们之间的基因数中最大的数和最小的数,算出它们之间的基因数中最大的数和最小的数,然后将它们之间的数作为向量,按两数中的大数排序至小数(即当大的数是位置靠后的数,则进行反向排序,否则正常排序),只有经逆转后,适应度值有提高的才接受下来,否则逆转无效
Reverse函数function SelCh=Reverse(SelCh,D) [row,col]=size(SelCh); ObjV=PathLength(D,SelCh); %计算路径长度 SelCh1=SelCh; for i=1:row r1=randsrc(1,1,[1:col]); r2=randsrc(1,1,[1:col]); mininverse=min([r1 r2]); maxinverse=max([r1 r2]); SelCh1(i,mininverse:maxinverse)=SelCh1(i,maxinverse:-1:mininverse); end ObjV1=PathLength(D,SelCh1); %计算路径长度 index=ObjV1<ObjV; SelCh(index,:)=SelCh1(index,:);
8 子代插入种群中
Chrom=Reins(Chrom,SelCh,ObjV);:主函数中的语句,调用Reins函数将子代插入种群中
Reins函数function Chrom=Reins(Chrom,SelCh,ObjV) NIND=size(Chrom,1); NSel=size(SelCh,1); [TobjV,index]=sort(ObjV); Chrom=[Chrom(index(1:NIND-NSel),:);SelCh];
解
最优解:
2—>1—>10—>9—>11—>8—>13—>7—>12—>6—>5—>4—>3—>14—>2
总距离:29.3405